/home/virtual/kjpt/journal//../xmls/kjpt-49-3-224.xml
한국산 도둑놈의갈고리족(콩과)의 DNA 바코드 및 계통학적 연구
적 요
본 연구에서는 DNA 바코드, 즉 엽록체 DNA의 rbcL, matK와 핵 리보솜 DNA의 ITS 염기서열을 이용하여 한국산 도둑놈의갈고리족 식물들의 종 식별을 실시하였다. 총 5개속 25분류군(총 75개체)의 식물에 대한 염기서열이 밝혀졌고, DNA 바코드 구간 조합에 따라 neighbor-joining 계통수들이 작성되었다. 그 결과, 종 식별률은 DNA 바코드 3개의 구간을 모두 사용하였을 때 가장 높게 나타났다(72%). rbcL+matK+ITS 계통수에서 본 족의 두 개의 아족과 다섯 속들은 단계통군으로 유집되었다. 도둑놈의갈고리아족군에서 도둑놈의 갈고리속과 갈고리속이 된장풀속보다 더 가깝게 묶였다. 갈고리속군에서 큰도둑놈의갈고리는 개도둑놈의갈고리 복합체의 자매군으로 형성되었고, 개도둑놈의갈고리의 종내분류군들은 각각 단계통을 형성하였다. 특히 형태적 변이로 인해 도둑놈의갈고리의 변종 또는 개도둑놈의갈고리와의 중간형으로 인식된 바 있는 긴도둑놈의갈고리는 개도둑놈의갈고리와 가장 가깝게 묶였다. 한편 도둑놈의갈고리의 변종이나 이명으로 인식되어온 애기도둑놈의갈고리는 변종으로 판단되었다. 싸리아족군의 경우, 매듭풀속 분류군들은 계통수상에서 각각 단계통으로 지지되었으나, 싸리속의 식물 16분류군 중 9분류군만 종식별이 가능하였다. 특히 싸리속내땅비수리절보다 싸리절에서 낮은 종판별 해상능이 나타났다(싸리절=28.5%, 비수리절=77.8%). 싸리속 식물 중에 비수리는 계통수상에서 형태적으로 유사한 자주비수리와 확연히 구분되었다. 잡종으로도 인식된 바 있는 해변싸리와 청비수리 중, 해변싸리는 독립종으로 판단되었다. 반면 청비수리는 잡종 여부를 판단할 수는 없었지만, 땅비수리와 근연 관계에 있는 것으로 사료된다.
주요어: 도둑놈의갈고리족, 종식별, DNA 바코드 rbcL, matK, ITS
Abstract
Species identification for the Korean tribe Desmodieae was conducted using the DNA barcoding genes rbcL, matK (from chloroplast DNA) and ITS (from nuclear ribosomal DNA). A total of 25 taxa (n = 75) in five genera were sequenced, and neighbor-joining trees were constructed using different combinations of DNA barcodes. When comparing these phylogenetic trees, a tree with all loci combined (rbcL + matK + ITS) showed the highest rate of identification success (72%). On this tree, two subtribes and five genera within the tribe were supported as monophyletic. In the Desmodiinae clade, Desmodium and Hylodesmum were more closely related to each other than to Ohwia. In the Hylodesmum clade, H. oldhamii was found to be a sister to H. podocarpum complex, and all taxa within the complex were identified successfully. Subsp. fallax, regarded as a variety of subsp. oxyphyllum, is closely clustered with subsp. podocarpum. Although var. mandshuricum has been regarded as a synonym of var. oxyphyllum, this taxon is supported as a distinct variety. For the Lespedezinae clade, all species of Kummerowia were monophyletic, while nine of 16 Lespedeza taxa were identified successfully. In particular, the resolution of Macrolespedeza (28.5%) was lower than that of Junceae (77.8%). Among the Lespedeza taxa, L. cuneata was distinguishable from L. lichiyuniae, despite morphological similarities. It has been suggested that both L. maritima and L. inschanica are hybrids. The former is thought to be an independent species. While it is difficult to determine whether the latter originated via hybridization, this study showed that it is closely related to L. juncea.
Keywords: Desmodieae, species identification, DNA barcode, rbcL, matK, ITS
DNA 바코드(DNA barcode)는 orthologous DNA 구간들의 짧은 염기서열을 이용하여, 형태적으로 식별하기 어렵거나 분류학적으로 문제가 있는 종들을 동정하는 기법이다( Kress et al., 2005). DNA 바코드는 식물이나 균류 등 다양한 분류군들의 동정과 계통학적 연구에 이용되고 있다( Purty and Chatterjee, 2016). 이상적인 DNA 바코드 구간은 근연종의 식별을 해결할 수 있을 정도로 충분히 가변적이고 저렴한 비용으로 실험을 할 수 있을 정도로 짧아야 하며, 여러 분류군들에 적용될 수 있어야한다( Dong et al., 2015). 최근 20여년간 각 생물군에 적용할 수 있는 이상적인 DNA 바코드 마커를 찾고자 많은 연구들이 수행되어 왔다( Hebert et al., 2003; Kress et al., 2005; CBOL Plant Working Group, 2009; Dong et al., 2012, 2015). 동물군의 경우에는 DNA 바코드 마커로 미토콘드리아의 cytochrome c oxidase I (COI) 유전자가 일반적으로 이용되고 있다( Kress and Erickson, 2008; Meusnier et al., 2008). 하지만 고등식물의 COI 유전자는 동물의 COI 유전자에 비해 진화속도가 느려 DNA 바코드 마커로 사용되기 어렵다( Kress et al., 2005). 이러한 이유로 식물군에서는 다른 DNA 마커들이 탐색되었고, 일반적으로 엽록체 DNA의 4개 구간( matK 유전자, rbcL 유전자, ycf1 유전자, psbA-trnH spacer)과 핵 ribosomal DNA의 internal transcribed spacer (ITS)가 마커로 추천되었다( Edwards et al., 2008; Hollingsworth et al., 2011; Dong et al., 2015; Vassou et al., 2015). 엽록체 DNA 중에서 rbcL과 matK 구간은 DNA barcode의 기본 마커로 쓰이는 구간이다( CBOL Plant Working Group., 2009). rbcL 구간은 polymerase chain reaction (PCR) 성공률이 높고 염기서열분석의 안정성이 좋으나, 종간 해상력이 떨어지는 단점이 있다( Renner, 1999; Salazar et al., 2003). 반대로 matK 구간은 PCR 성공률과 시퀀싱의 안정성은 상대적으로 떨어지나 종식별률이 높은 것으로 알려져 있다( Hollingsworth, 2011). ycf1 구간의 경우에는 높은 염기서열 변이를 보이지만 이로 인해 primer를 제작하는 데 어려움이 있다( Dong et al., 2015). psbA-trnH 구간은 non-coding 구간으로 길이가 짧으며 시퀀싱 안정성이 높고 rbcL이나 matK와 조합하였을 때 일부 분류군에서 높은 종식별률을 보였다( Vassou et al., 2015). ITS 구간은 식물의 분자계통학적 연구 및 DNA 바코드 연구에 가장 많이 이용되는 구간 중 하나로(e.g., Riahi et al., 2011; Youm et al., 2016; Choi et al., 2018; Jabbour et al., 2018), 엽록체 DNA에 비해 진화속도가 빠르기 때문에 구간의 길이에 비해 계통학적으로 유용한 염기서열의 비율이 많은 것으로 알려져 있다( Wolfe et al., 1987). 도둑놈의갈고리족(tribe Desmodieae)은 콩과(family Fabaceae)에 속하고 32속 530여종으로 이루어진 것으로 알려져 있으나( Ohashi, 2005), 최근 형태 및 분자계통학적 연구 결과에 따르면 39속으로 인식된 바 있다( Ohashi et al., 2018a, 2018b). 본 족의 식물들은 초본부터 교목까지 다양한 생활형을 보이고( Ohashi, 2005), 이들은 주로 열대, 아열대, 온대지역에서 분포하나 일부 종들은 동아시아와 북아메리카의 한대와 아한대 지역에서 자란다( Ohashi, 2005). 도둑놈의갈고리족은 열매의 형태와 엽병 아래에 달리는 탁엽의 유무, 기판 기부에 귀모양 부속체(auricle)의 유무 등으로 도둑놈의갈고리아족(subtribe Desmodiinae)과 싸리아족(subtribe Lespedezinae)으로 구별되는 것으로 알려져 있다( Ohashi et al., 1981; Jabbour et al., 2018). 도둑놈의갈고리족 식물들은 사료, 토양의 비옥화, 사면안정화 등을 목적으로 심어지고, 일부 종들은 약용으로 사용되기도 하여( Ohashi, 2005), 식물자원으로 가치가 높다. 본 족의 식물들을 포함한 콩과 식물의 분자계통학적 연구는 여러 차례 수행된 바 있다( Kajita et al., 2001; Legume Phylogeny Working Group, 2013, 2017). 이러한 분자계통학적 연구 결과들은 본 족이 콩아과(Papilionoideae) 내에 단계통임을 잘 지지했으며, 두 개의 아족들도 각각 단계통을 형성하였다. 최근 도둑놈의갈고리족의 많은 속이 포함된 분자계통학적 연구( Jabbour et al., 2018)에서 속간 유연관계와 분류체계에 대해 논의된 바 있다. 싸리아족의 경우, 싸리속( Lespedeza Michx.)을 중심으로 형태분류학적 연구( Lee, 1965; Hatusima, 1967; Akiyama, 1988)가 수행되었고, 최근 분자계통학적 연구는 싸리속내 종간 유연관계 및 근연속들과의 유연관계 등에 대해 분석하였다( Han et al., 2010; Nemoto et al., 2010; Xu et al., 2012). 한편 도둑놈의갈고리아족은 도둑놈의갈고리속( Desmodium Desv.)을 중심으로 분류학 및 계통학적 연구들이 수행되어 왔다( Ohashi, 1973; Kajita and Ohashi, 1994; Kajita et al., 2001; Jabbour et al., 2018; Ohashi et al., 2018a, 2018b). 도둑놈의갈고리속은 계통수상에서 다계통으로 확인되어왔는데, 이를 반영하여 현재 본 속내 많은 분류군들이 새로운 속으로 분류되었다(e.g., Ohashi, 1999; Ohashi and Mill, 2000; Ohashi and Ohashi, 2012, 2018a, 2018b; Ohashi et al., 2018a, 2018b). 한반도에는 2아족 6속의 도둑놈의갈고리족 식물 즉 도둑놈의갈고리아족 내에 도둑놈의갈고리속, 갈고리속( Hylodesmum H. Ohashi & R. R. Mill), 된장풀속( Ohwia H. Ohashi) 3속, 싸리아족 내에 꽃싸리속( Campylotropis Bunge), 매듭풀속( Kummerowia Schindl.), 싸리속 3속이 분포하는 것으로 알려져 있다( Choi, 2007) ( Fig. 1). 한편 최근 잔디갈고리의 학명은 Desmodium heterocarpon (L.) DC.에서 Grona heterocarpa (L.) H. Ohashi & K. Ohashi로 변경된 바 있다( Ohashi and Ohashi, 2018a). 이들 중 싸리속과 갈고리속은 상대적으로 많은 분류군들을 포함하고 있으며 종간 분류에 어려움이 있다. 한국에 자라는 싸리속 식물들은 땅비수리절[section Junceae (Maxim.) H. Ohashi & T. Nemoto]과 싸리절(section Macrolespedeza Maxim.)로 구분되는데( Ohashi and Nemoto, 2014), 땅비수리절 식물들은 개방화와 폐쇄화가 모두 달리는데 비해 싸리절 식물들은 개방화만 달리는 특징으로 구분된다. 싸리절 식물들은 주로 녹화용으로 쓰이며, 땅비수리절 식물들은 비료나 거름 등으로 쓰인다. 땅비수리절에 속하는 비수리[ L. cuneata (Dum. Cours.) G. Don]는 야관문이라는 약용명으로도 알려져 있는데, 최근 민간의 관심을 받으며 식품이나 약재자원으로 연구되고 있다( Chung and Cheong, 2016). 하지만 본 속의 식물들은 큰 형태적 변이폭을 보이며 간혹 종간 한계가 명확하지 않다. 이러한 다양한 형태적 변이는 학자에 따라 많은 종내분류군이나 독립종으로 인식되었고( Nakai, 1927; Lee, 1965, 1980; Hatusima, 1967; Akiyama, 1988), 청비수리[ L. inschanica (Maxim.) Schindl.]와 해변싸리( L. maritima Nakai) 등이 잡종으로 처리되었다( Lee, 1965, 1980). 따라서 외부형태에 따라 본 속의 식물들을 동정하는 것은 어려운 것으로 알려져 있다. 한편, 국내 갈고리속 식물들은 된장풀[ O. caudata (Thunb.) H. Ohashi]과 함께 도둑놈의갈고리속으로 여겨져 왔으나( Choi, 2007), 영주갈고리[ H. laxum (DC.) H. Ohashi & R. R. Mill subsp. laxum]의 한국내 분포가 처음으로 보고되면서 갈고리속으로 분리된 바 있다( Lee et al., 2012). 갈고리속 식물들 중에 큰도둑놈의갈고리[ H. oldhamii (Oliv.) H. Ohashi & R. R. Mill]는 다른 식물들에 비해 많은 소엽(5–7)을 가지는 점( Choi, 1991, 2007), 영주갈고리는 잎상면이 빛나며 다소 상록성을 띄며, 열매에 종자가 3개씩 달리는 특징으로 뚜렷하게 구분된다( Lee et al., 2012). 하지만 개도둑놈의갈고리 복합체[ H. podocarpum (DC.) H. Ohashi & R. R. Mill complex]의 종내분류군 즉 개도둑놈의갈고리(subsp. podocarpum), 긴도둑놈의갈고리[subsp. fallax (Schindl.) H. Ohashi & R. R. Mill], 도둑놈의갈고리[subsp. oxyphyllum (DC.) H. Ohashi & R. R. Mill var. oxyphyllum], 애기도둑놈의갈고리[subsp. oxyphyllum var. mandshuricum (Maxim.) H. Ohashi & R. R. Mill]는 일부 개체들에서 심한 형태적 변이를 보이고 있다( Choi, 1991). 또한 엽록체 유전체의 제한효소 절편길이 다형성 분석에 기반한 계통학적 연구에서 개도둑놈의갈고리 종내분류군간 한계가 뚜렷하지 않은 것으로 나타났다( Kajita and Ohashi, 1994). 따라서 본 연구는 한국산 도둑놈의갈고리족 식물들을 대상으로 DNA 바코드를 이용한 종식별과 이들의 계통학적 유연관계 대해서 논의하고자 하였다.
재료 및 방법
재료 수집
본 연구에 사용된 도둑놈의갈고리족 식물은 국내에 자생하고 있는 종을 대상으로 선정하여 총 25종 75개체가 수집되었다( Appendix 1). 수집된 잎은 신선한 잎을 선별하여 DNA 추출을 위해 실리카겔에 건조시켰다. 본 연구에서 포함된 분류군들은 개체 및 집단 간 염기서열에 의한 차이와 무관하게 계통수상에서 분류군 별로 동정이 가능한지 보기 위해, 분류군 당 3개체씩 서로 다른 곳에서 수집하였다. 일부 같은 장소에서 채집된 분류군들의 경우에는 10 m 이상 거리를 띄어 같은 개체가 채집되는 것을 피했다. 채집된 재료는 국립생물자원관 관속식물표본 수장고(KB)와 인하대학교 식물표본관(IUI)에 수장하였다.
DNA 추출 및 PCR
염기서열 분석을 위해 채집된 식물들의 건조된 잎에서 DNA를 추출하였으며, DNA 추출에는 DNeasy Plant Mini Kit (Qiagen, Seoul, Korea)를 이용하였다. 추출된 DNA는 2% agarose gel에 전기영동하여 유무와 상태를 확인하였고, DNA의 농도를 NanoDrop ND-1000 (NanoDrop Technologies, Wilmington, DE, USA)을 사용하여 측정하였다. 본 연구에서는 총 3구간( rbcL, matK, ITS)의 염기서열 정보를 얻고자, 추출된 DNA들을 대상으로 PCR을 수행하였다. Primer는 기존의 연구들 및 본 연구에서 새롭게 개발한 primer를 사용하였다( Appendix 2). ITS 구간을 증폭하기 위해서 primer ITS5와 ITS4가 쓰였고( White et al., 1990), matK 구간을 증폭하기 위해 2쌍의 primer ( matK1100L, trnK2R; matK4La, matK1932)가 사용되었다( Hasebe et al. 1994; Wojciechowski et al., 2004). 하지만 일부 분류군에서 matK4La와 matK1932를 이용한 PCR 성공률이 매우 낮아, FGA309F와 FGA309R를 새롭게 제작하여 적용하였다. rbcL 구간을 분석할 때는 rbcLaF ( Hasebe et al., 1994)의 reverse primer인 rbcLcr ( Hasebe et al., 1994)이 일부 분류군에 적용되지 않아, rbcLafR을 사용하였다. PCR 반응용액의 조성은 DNA 10–20 ng (1 μL), Dr. Taq Master Mix without dye (2×) 10 μL (0.4 mM dNTPs, 2× PCR buffer with 4 mM MgSO 4, and 0.4 U/μL; MG- med, Seoul, Korea) DNA polymerase, 0.3 μM의 forward, reverse primer를 각각 0.5 μL씩 넣어 총 볼륨 20 μL가 되도록 하였다. PCR 조건은 95oC에서 3분간 pre-denaturation을 진행한 후, 95oC에서 30초 동안 denaturation, 55oC에서 1분 동안 annealing, 72oC에서 extension을 실시하였고, ITS구간은 45초, matK, rbcL 구간은 90초로 구성된 반응을 총 35회 반복하고 final extension을 72oC에서 7분동안 진행하였다. 이후 2% agarose gel에서 PCR 반응 여부를 확인한 후, 성공적으로 증폭된 개체들은 Macrogen Inc. (Seoul, Korea)에 염기서열 분석을 의뢰하였다. 분석장비로는 ABI 3730XL sequencer (Applied Biosystems, Foster City, CA, USA)가 사용되었다.
자료 분석
본 연구에서 분석된 염기서열들의 Chromatogram을 Geneious R 7.1.9 (Biomatters Ltd., Auckland, New Zealand)를 이용하여 결정하였고 GenBank에 등록하였다(accession no. MK933477–MK933701). 최종 결정된 염기서열들을 MUSCLE ( Edgar, 2004)을 사용하여 정렬하였다. 위의 염기서열 정보를 이용하여 한국산 도둑놈의갈고리족 식물들의 Neighbor-Joining (NJ) 계통수가 작성되었고, 이를 위해 PAUP ( Swofford, 2002)을 이용하였다. NJ 계통수 작성을 위한 개체간 유전적 거리(genetic distance)는 Kimura’s two parameter model ( Kimura, 1980)을 기반으로 계산하였다. 계통수의 각 마디에 대한 지지도는 bootstrap value (BV)로 나타냈고, 2,000 반복과정을 거쳐 PAUP을 통해 측정되었다. 계통 분석의 군외군(Outgroup)으로 두 종이 선택되었다. 한 종은 도둑놈의갈고리족과 같은 Phaseoloid group에 속하는 콩[Glycine max (L.) Merr.] ( rbcL and matK, NC_007942; ITS, FJ609734)이고( Legume Phylogeny Working Group, 2013), 다른 한 종은 본 족과 다른 아과(subfamily Mimosoideae)에 속하는 Acacia ligulata Aiton ex Steud. ( rbcL and matK, NC_026134; ITS, KC200918)이다( Legume Phylogeny Working Group, 2017). 결과적으로 3개 구간의 염기서열자료를 각각 독립적으로 수행하거나 가능한 조합으로 유합하여 총 7개의 자료행렬을 구성하여 분석하였다. 종식별 해상력은 작성된 NJ 계통수를 확인하여 전체 종 중 단계통으로 유집된 종의 비율을 통해 구했다. 한편 앞서 측정된 염기서열 간 유전적거리를 이용해 종내·간 유전자 구간들이 바코드 갭(barcode gap)을 나타내는지 확인하였다. 여기서 바코드 갭이란 측정된 종내 유전적 거리의 범위와 종간 유전적 거리의 범위가 겹치지 않는 경우를 뜻한다( Meyer and Paulay, 2005). 또한 염기서열의 변이율은 maximum parsimony 분석을 통해 확인하였다.
결 과
이번 연구를 통해 한국산 도둑놈의갈고리족(Desmodieae) 식물, 총 25분류군 75개체의 rbcL, matK, ITS 구간 염기서열이 밝혀졌다. 최종적으로 DNA 바코드 구간의 염기서열 분석 성공률(sequencing success)은 100%로 높게 나타났다( Table 1). 도둑놈의갈고리아족(Desmodiinae) 개체들은 matK4La와 matK1932Ra에 의해 증폭되는 구간에서 PCR 성공률이 매우 떨어졌지만, 새롭게 제작된 primer인 FGA309F와 FGA309R를 통해 모두 분석되었다( Appendix 2). 염기서열 분석 결과, rbcL의 길이는 715 bp로 일정하였고, matK와 ITS는 각각 813–822 bp, 594–648 bp로 변이를 보였다. 염기서열 내 변이율은 ITS (13.40%), matK (12.98%), rbcL (3.22%)로 높았다( Table 1). Kimura’s two parameter에 근거한 종내 유전적거리에서 ITS (0.0026)가 가장 높은 평균값을 보였고, 그 다음은 matK (0.0019), rbcL (0.0002) 순이었다( Table 2). 종간 유전적 거리에서도 ITS 구간이 0.1099로 확연히 높았고, matK (0.0179)와 rbcL (0.0192)은 비슷한 수치를 보였다( Table 2). 본 연구대상 식물들의 바코드 갭의 여부를 확인해본 결과, 0–0.5 구간에서 종내, 종간 유전적 거리가 겹쳐져 본 연구대상종간 확연한 바코드 갭은 없었다. 본 연구에서 분석된 구간들( rbcL, matK, ITS)을 다양하게 조합하여 한국산 도둑놈의갈고리족 식물에 대한 NJ 계통수들이 작성되었다. 대체로 단일구간의 염기서열을 이용했을 때( matK=20%, rbcL=28%, ITS=68%)보다는 두 구간 이상을 조합하였을 때 종식별률이 높은 것으로 나타났고( matK+ rbcL=32%, matK+ITS=64%, rbcL+ITS=68%; all=72%), 특히 ITS 구간이 포함될 때 더 높은 해상능을 보였다. 이들 중 엽록체 DNA ( rbcL+ matK), ITS, 전체 구간 결과를 이용한 세개의 계통수들을 각각 Figs. 2– 4에 나타냈다. 이러한 계통수들은 분류군간 유연관계에서 차이를 보였다. 먼저 3구간 유합( rbcL+ matK+ITS)계통수와 ITS계통수는 본 족에 속하는 두개의 아족을 모두 단계통으로 지지하였으나( Figs. 3, 4), 엽록체 DNA 계통수에서 도둑놈의갈고리아족은 다계통으로 나타났다( Fig. 2). 속간 유연관계에서 3구간 유합 계통수와 엽록체 DNA 계통수는 모든 속을 단계통으로 지지하였으나, ITS 계통수는 싸리속( Lespedeza)을 단계통으로 지지하지 않았다( Fig. 3). 3구간 유합 계통수와 엽록체 DNA 계통수간 차이도 확인되었는데, 전자에서 도둑놈의갈고리속( Desmodium)과 갈고리속( Hylodesmum)이 된장풀속( Ohwia)에 비해 가깝게 유집되었지만( Figs. 3, 4), 엽록체 DNA 계통수는 갈고리속과 된장풀속이 더 가깝게 묶였다( Fig. 2). 또한 3구간 유합 계통수에서 싸리절( Macrolespedeza)은 별개의 분계조를 형성하였지만, 엽록체 DNA 계통수에서는 두개의 절이 별개로 구분되지 않았다. 한편 종식별 결과를 보면, 다른 계통수들에서 단계통으로 지지된 분류군들은 3구간 유합 계통수에서도 모두 단계통으로 나타났지만, 조록싸리( L. maximowiczii)는 ITS 계통수상에서만 단계통으로 지지되었다. 엽록체 DNA 계통수(32%)는 ITS (68%)나 3구간 유합 계통수(72%)에 비해 종식별률이 낮았는데, 특히 싸리속에서 좀싸리[ L. virgata (Thunb.) DC.]를 제외하면 단계통으로 지지되는 분류군은 없었다. 한국산 도둑놈의갈고리족 식물들에 대해 가장 높은 종식별률을 보인 DNA 바코드 3구간 유합( rbcL+ matK+ITS) 계통수를 기준으로 종간 유연관계를 보았다( Fig. 4). 먼저 도둑놈의갈고리아족의 모든 분류군은 단계통을 형성하여 100%의 종식별률을 보였다. 본 계통군에서 된장풀(O. caudata)이 가장 먼저 분지되었고 그 다음 잔디갈고리( D. heterocarpon)와 갈고리속 분류군들이 서로 자매군으로 나타났다. 갈고리속 계통군에서는 큰도둑놈의갈고리( H. oldhamii)가 가장 먼저 분지되었고, 나머지 개도둑놈의갈고리 복합체( H. podocarpum complex) 4분류군은 각각 분류군별로 계통군을 형성하였다. 개도둑놈의갈고리 복합체 중, 같은 아종내에서 변종수준으로 구분되는 도둑놈의갈고리(subsp. oxyphyllum var. oxyphyllum)와 애기도둑놈의갈고리(subsp. oxyphyllum var. mandshuricum)는 다른 아종들에 비해 서로 가깝게 유집되었다. 한편 싸리아족 계통군에서 두 속 또한 모두 단계통으로 구분되었다. 먼저 매듭풀속(Kummerowia)의 경우 매듭풀[K. striata (Thunb.) Schindl.]과 둥근매듭풀[K. stipulacea (Maxim.) Makino] 모두 단계통을 형성하였다. 싸리속 계통군에서 싸리절은 하나의 분계조로 유집되었으나(BV=100%), 땅비수리절(Junceae)은 다계통으로 확인되었다. 본 속의 종식별 결과를 보면, 싸리속의 9종 즉 해변싸리(L. maritima), 큰잎싸리(L. davidii Franch.), 호비수리[L. davurica (Laxm.) Schindl.], 비수리(L. cuneata), 자주비수리(L. lichiyuniae T. Nemoto, H. Ohashi & T. Itoh), 괭이싸리[L. pilosa (Thunb.) Siebold & Zucc.], 분홍싸리(L. floribunda Bunge), 개싸리[L. tomentosa (Thunb.) Siebold ex Maxim.], 좀싸리는 단계통으로 유집되었다. 하지만 나머지 7종 즉 싸리(L. bicolor Turcz.), 참싸리(L. cyrtobotrya Miq.), 검나무싸리(L. melanantha Nakai), 풀싸리[L. thunbergii (DC.) Nakai subsp. thunbergii], 조록싸리(L. maximowiczii C.K. Schneid.), 땅비수리[L. juncea (L. f.) Pers.], 청비수리(L. inschanica)는 단계통으로 묶이지 않았다. 이러한 결과를 종합하였을 때, 싸리속내 비수리절과 싸리절은 각각 77.8% 및 28.5%의 종식별률을 보였다.
고 찰
본 연구에서는 rbcL, matK, ITS 구간 염기서열을 사용한 결과 72%로 한국산 도둑놈의갈고리족(Desmodieae)의 식물들을 동정할 수 있었다( Fig. 4, Table 3). 이러한 결과는 ITS에 기반한 DNA 바코드를 이용한 콩과 식물들에 대한 종식별 결과와 비슷한 수치로 나타났고( Gao et al., 2010), 엽록체 DNA의 두 구간을 이용한 종식별률(32%)에 비해 매우 높은 값이었다( Table 3). 3구간을 DNA 바코드로 사용한 결과(72%)는 ITS만을 사용했을 때보다 높은 종식별률(68%)을 보였는데, 이는 엽록체 DNA 두 구간을 포함함으로써 염기서열 내 변이 수가 늘어났기 때문으로 생각된다( Table 1). 한편 바코드 내에 분석 비용을 고려할 때는 ITS만 이용하는 것이 경제적인 것으로 판단된다( Fig. 3, Table 3). 하지만 비수리( Lespedeza cuneata)를 자주비수리( L. lichiyuniae)로부터 구별된다는 점에서 땅비수리절( Junceae) 식물들을 식별할 때는 엽록체 DNA 두 구간을 포함하는 것을 고려할 수 있다. 엽록체 DNA를 이용하였을 때보다 ITS에서 높은 종식별률이 나타나는 이유는 ITS 구간의 높은 염기서열 변이율 때문으로 보인다( Table 1). 한편, DNA 바코드들은 속 수준에서 동정하는 데 있어서는 DNA 바코드 조합과 상관없이 100%의 높은 해상력을 보였다. 속내 종 동정에서 싸리속( Lespedeza) 일부 분류군들은 단계통으로 지지되지 않아 바코드를 이용한 종식별에 문제가 있는데, 본 속 식물들을 제외했을 때 종식별률은 100%로 높아졌다. 종식별 결과를 종합하였을 때, 한국산 도둑놈의갈고리족의 대부분 식물들에 대한 종식별은 본 연구에서 사용된 3 구간( rbcL, matK, ITS)으로 대부분 가능한 것으로 판단된다. 본 연구에서 쓰인 DNA 바코드들은 염기서열 분석 성공률(100%)이 높아 효용성이 높은 것으로 판단된다( Table 1). DNA 바코드 3구간 유합 자료( rbcL+ matK+ITS)를 이용한 계통학적 분석 결과를 바탕으로 두개의 아족들에 대해 논의하였다( Fig. 4). 먼저 도둑놈의갈고리아족(Desmodiinae) 내 3개 속 즉 된장풀속( Ohwia), 도둑놈의갈고리속( Desmodium), 갈고리속( Hylodesmum)의 유연관계를 확인하였을 때, 강하게 지지되지 않았지만 도둑놈의갈고리속과 갈고리속이 더 가깝게 연관되는 것으로 나타났다(BV=54.7%). Ohashi (2005)는 rbcL 계통수와 외부형질을 근거로 도둑놈의갈고리아족내 속들을 두개의 그룹으로 분류한 바 있다. 하나는 Phyllodium 그룹으로 된장풀속 등을 포함하고 다른 그룹은 Desmodium 그룹으로 도둑놈의갈고리속과 갈고리속 등으로 구성된다. 본 연구결과에서 된장풀( O. caudata)군이 잔디갈고리와 갈고리속 분류군들의 자매군으로 나타나, Ohashi(2005)의 견해를 지지하였다. 큰도둑놈의갈고리( H. oldhamii)는 개도둑놈의갈고리( H. podocarpum subsp. podocarpum)가 포함된 복합체들과 달리 3출엽이 아닌 5엽 우상복엽을 가지며 Pinnata 아절로 분류되는데( Ohashi, 1973; Kajita and Ohashi, 1994), 본 계통수상에서도 갈고리속 식물 중 가장 먼저 분지되었다. 한편 엽록체 DNA 단편 길이를 이용한 개도둑놈의갈고리 복합체의 계통학적 연구에서 한국에 분포하는 4 분류군 즉 도둑놈의갈고리( H. podocarpum subsp. oxyphyllum var. oxyphyllum)와 애기도둑놈의갈고리( H. podocarpum subsp. oxyphyllum var. mandshuricum), 개도둑놈의갈고리, 긴도둑놈의갈고리( H. podocarpum subsp. fallax)는 낮은 BV로 인해 관계를 파악할 수 없었다( Kajita and Ohashi, 1994). 하지만 본 연구 결과에서 개도둑놈의갈고리 복합체의 분류군들은 모두 단계통으로 지지되었고, 긴도둑놈의갈고리는 도둑놈의갈고리보다는 개도둑놈의갈고리와 가깝게 유집되었다. 이러한 결과는 ITS 및 rps16- trnQ, trnL-F를 이용한 분석결과와 일부 일치하였다( Li et al., 2019). 도둑놈의갈고리족 식물들의 유연관계를 나타내는데 있어서 엽록체 DNA 단편 길이 분석보다는 염기서열 자료가 더 높은 해상력을 보이는 것으로 생각된다. 긴도둑놈의갈고리는 도둑놈의갈고리와 개도둑놈의갈고리의 중간적인 형태를 가지는 것으로 처음에 인식되었고( Schindler, 1916), 이후 도둑놈의갈고리의 변종으로 인식되기도 하였다( Ohashi, 1973). Choi (1991)는 잎 형태 관찰 결과에서 각 분류군들의 소엽의 형태가 뚜렷이 구분되는 것을 근거로 이들을 별개 분류군으로 인식하였다. 본 계통수의 결과도 이러한 소엽의 형태에 근거한 분류학적 견해를 지지하였다. 애기도둑놈의갈고리는 도둑놈의갈고리와 유사한 형태를 띄지만 잎이 식물체의 중간부에 모여 나는 것을 근거로 도둑놈의갈고리의 변종으로 여겨지는데( Ohashi, 1973), 학자에 따라서는 애기도둑놈의갈고리를 도둑놈의갈고리의 이명으로 취급하기도 한다( Huang and Ohashi, 2010). 이번 연구의 계통수상에서 애기도둑놈의 갈고리는 도둑놈의갈고리와 가장 가까운 근연관계를 보였다. 또한 도둑놈의갈고리 한 개체의 채집지와 애기도둑놈의갈고리 개체들의 채집지가 매우 가까움에도 불구하고 두 분류군이 유전적으로 구분되었다. 하지만 두 분류군이 식물체에서 잎이 달리는 위치가 차이나는 점을 제외하면 형태적으로 연속적인 변이를 보이는 점 등을 볼 때, 애기도둑놈의갈고리는 도둑놈의갈고리의 변종 수준으로 인식하는 것이 타당할 것으로 생각된다. 싸리아족(Lespedezinae)군에서 싸리속과 매듭풀속( Kummerowia)은 각각 단계통을 형성하여 기존의 분자계통학 연구결과와 일치하였다( Han et al., 2010). 매듭풀속 두 종 모두 별개의 분계조로 나뉘었다. 하지만 싸리속의 땅비수리절은 다계통으로 나타났다. 한편 본 속의 16분류군중에서 단계통을 형성하지 못한 분류군들은 7분류군 즉 땅비수리( L. juncea), 청비수리( L. inschanica), 싸리( L. bicolor), 풀싸리( L. thunbergii), 검나무싸리( L. melanantha), 참싸리( L. cyrtobotrya), 조록싸리( L. maximowiczii)로 확인되었다. 땅비수리절 식물들은 DNA 바코드를 이용하였을 때 77.8%의 종식별률을 보여 비교적 잘 구분되었다. 단계통을 형성하지 않은 분류군 중, 청비수리는 땅비수리와 유사하지만, 식물체 아래부분에 달리는 잎은 호비수리( L. davurica)처럼 넓어지는 것을 특징으로 땅비수리와 호비수리의 잡종[ Lespedeza× divaricata (Nakai) T. Lee]으로 여겨졌다( Lee, 1965; Choi, 2007). 본 연구의 엽록체 두 구간( rbcL+ matK)의 계통수에서 청비수리는 근연의 두 종과 유연관계를 파악할 수 없었다. 반면 ITS가 포함된 계통수에서는 청비수리, 땅비수리, 호비수리가 하나의 분계조를 형성하였다. 본 연구결과는 청비수리의 잡종 기원에 대해 논의하기에 다소 해상력이 떨어지지만, 호비수리만 단계통을 형성하는 점에서 청비수리는 근연두 종간의 잡종이라기보다는 땅비수리와 가까운 유연관계를 갖는 것으로 생각된다. 자주비수리는 최근에 국내에서 분포가 알려진 식물로 전체적인 형태는 비수리와 유사하지만, 꽃이 자주색이고 잎 뒷면과 열매에 털이 많은 점에서 구분된다( Han and Choi, 2008). 최근에 비수리의 면역력 향상과 항산화 효과 등이 알려지며 많은 관심을 받고 있다( Eun, 2011; Kim et al. 2011). 따라서 본 종을 이용하기 위해서는 근연분류군들과의 종식별이 중요하다. 현재 연구결과는 자주비수리와 비수리의 가까운 유연관계를 나타냈고, 두 분류군을 각각 단계통으로 지지하였다. 따라서 한국산 비수리와 자주비수리는 본 연구에서 사용된 구간들을 이용하여 식별 가능하다. 한편 싸리절 식물들은 땅비수리절에 비해 더 낮은 종식별률을 보였고(28.5%), 이전 싸리속의 계통학적 연구결과와 일치하였다( Xu et al., 2012, 2017). 이러한 결과는 싸리절 식물의 진화에서 잡종화 현상이 중요한 역할을 했기 때문에 비롯된 것으로 생각된다( Xu et al., 2017). 본 연구결과에서 단계통을 형성하지 않은 5종들(조록싸리, 참싸리, 싸리, 검나무싸리, 풀싸리)은 심한 형태적 변이를 보이는 개체들로 인해 오동정 되는 경우가 많다. 반면 단계통으로 지지된 2종 즉 해변싸리( L. maritima)와 큰잎싸리( L. davidii)는 독특한 형태 및 분포 특징을 보인다. 먼저 해변싸리는 잎 상면에 광택이 있고 혁질이며, 엽연은 비후되거나 말린다( Lee, 1980; Choi, 2007). 싸리절 대부분 식물들이 한·중·일에 걸쳐 넓게 분포하는데 비해 해변싸리는 한국 남부지역의 해안 산지에만 자란다( Choi, 2007). 또한 개화기는 9월에서 10월로 다른 종들에 비해 늦는 것으로 알려져 있다. 본 종은 이전 계통학적 연구들( Han et al., 2010; Nemoto et al., 2010; Xu et al., 2012)에서 포함되지 않아 근연분류군들과의 관계 및 단계통 형성 여부를 파악할 수 없었는데, 이러한 형태 및 지리적 차이 등을 고려할 때, 해변싸리는 비교적 다른 종들에 비해 종분화가 진행된 것으로 사료된다. 한편 해변싸리는 형태적 특징을 기초로 Lee ( 1965, 1980)에 의해 근연분류군인 조록싸리와 참싸리의 잡종으로 여겨졌으나, 본 연구에서 본 종이 단계통으로 지지되었고 부모로 생각된 두 종과 가까운 관계를 보이지 않았으므로 이러한 가설을 지지하지 않는 것으로 판단된다. 큰잎싸리는 소엽이 다른 종에 비해 가장 크며[3.5–7(–13)×2.5–5(–8) cm] 식물체 전체에 퍼진 털이 밀생한다( Huang et al., 2010). 본 종은 ITS와 phosphoglycerate kinase (PGK)를 이용한 계통학적 연구( Xu et al., 2017)에서 단계통으로 유집되지 않았지만, 본 연구에서 두 종 모두 나머지 한국산 싸리들과 구분되었다. 이러한 결과는 한국산 큰잎싸리들은 최근 도로공사 등으로 인해 자생지인 중국에서 도입되었기 때문으로 생각된다( Han and Choi, 2008). 본 연구를 통해 한국산 도둑놈의갈고리족을 식별하기 위한 바코드가 평가되었고, 이들의 계통학적 유연관계에 대해 고찰하였다. 대부분의 분류군은 식별 가능하였고, 계통학적 유연관계 또한 기존의 분류학적 체계를 지지하였다. 하지만 싸리속 일부 분류군은 종식별률이 낮게 나타났는데, 새로운 분자마커 탐색이 필요해 보인다.
ACKNOWLEDGMENTS
We thank I.S. Choi of University of Texas at Austin for his help to analyze data and comment on this manuscript. The authors also appreciate to C.E. Lim of the National Institute of Biological Resources (NIBR) for providing samples. This work was supported by a grant (No. NBIR201812102) from NIBR.
Fig 1.
Six genera of tribe Desmodieae in Korea. A. Desmodium heterocarpon, photo by W. B. Cho, 29 Aug 2009, Sallok road, Harye-ri, Jejudo. B. Hylodesmum podocarpum subsp. podocarpum, photo by D. P. Jin, 30 Sep 2016, Isl. Oeyeon, Chungcheongnam-do. C. Ohwia caudata, photo by J. S. Park, 21 Aug 2017, Dongbaekdongsan, Jeju-do. D. Campylotropis macrocarpa, photo by D. P. Jin, 15 Sep 2017, Paljoryeong, Cheongdo-gun, Gyeongsangbuk-do. E. Kummerowia striata, photo by D. P. Jin, 4 Aug 2017, Buldang-ri, Gwangju-si, Gyeonggi-do. F. Lespedeza cuneata, photo by J. S. Park, 10 Aug 2018, Sorae Marsh Ecological Park, Incheon-si. 
Fig 2.
The neighbor-joining phylogenetic tree of the Korean tribe Desmodieae based on combined sequence from rbcL and matK. The number from node indicate bootstrap value (>50%). 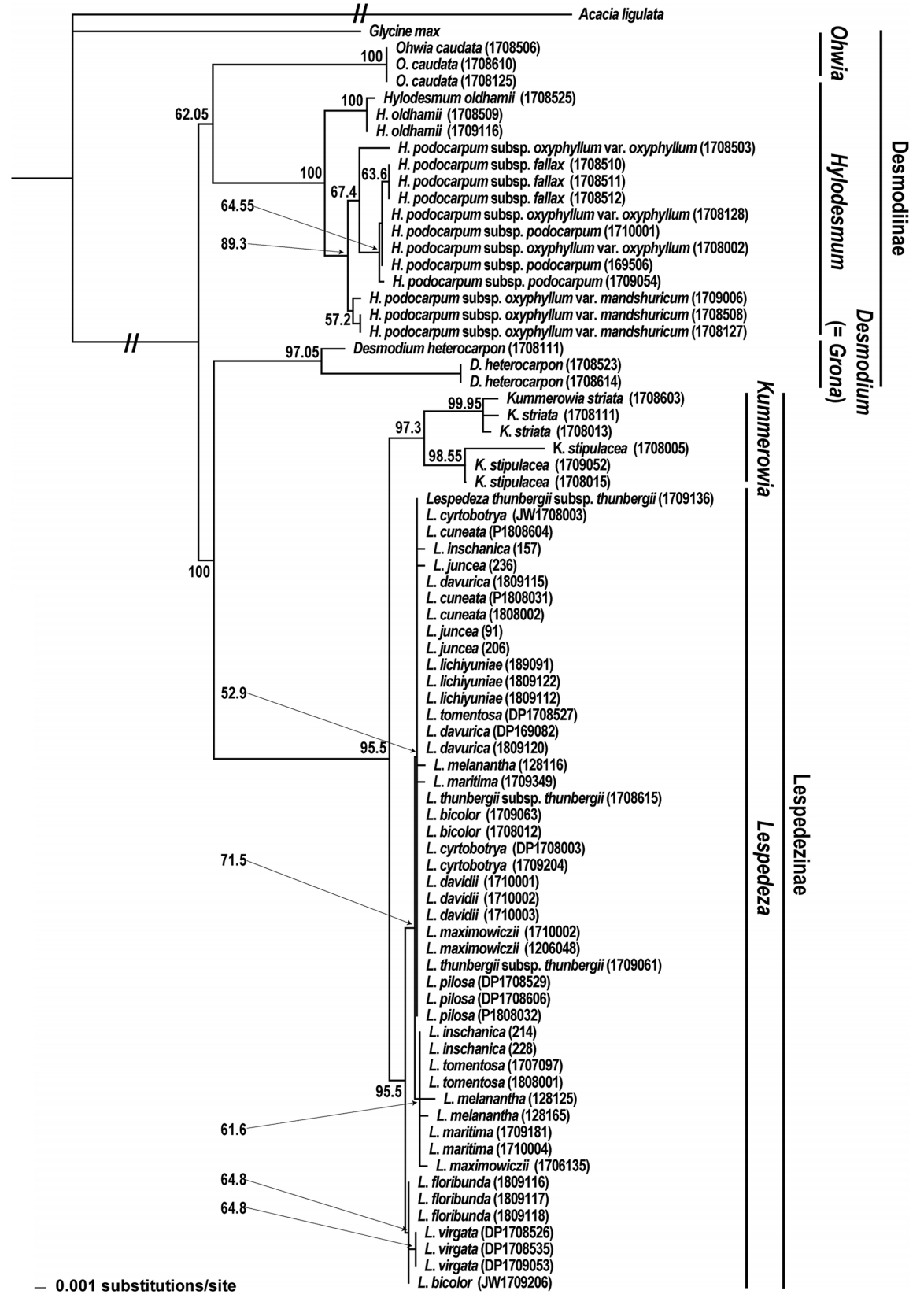
Fig 3.
The neighbor-joining phylogenetic tree of the Korean tribe Desmodieae based on internal transcribed spacer. The number from node indicate bootstrap value (>50%). 
Fig 4.
The neighbor-joining phylogenetic tree of the Korean tribe Desmodieae based on combined sequence from rbcL, matk, and internal transcribed spacer. The number from node indicate bootstrap value (>50%). 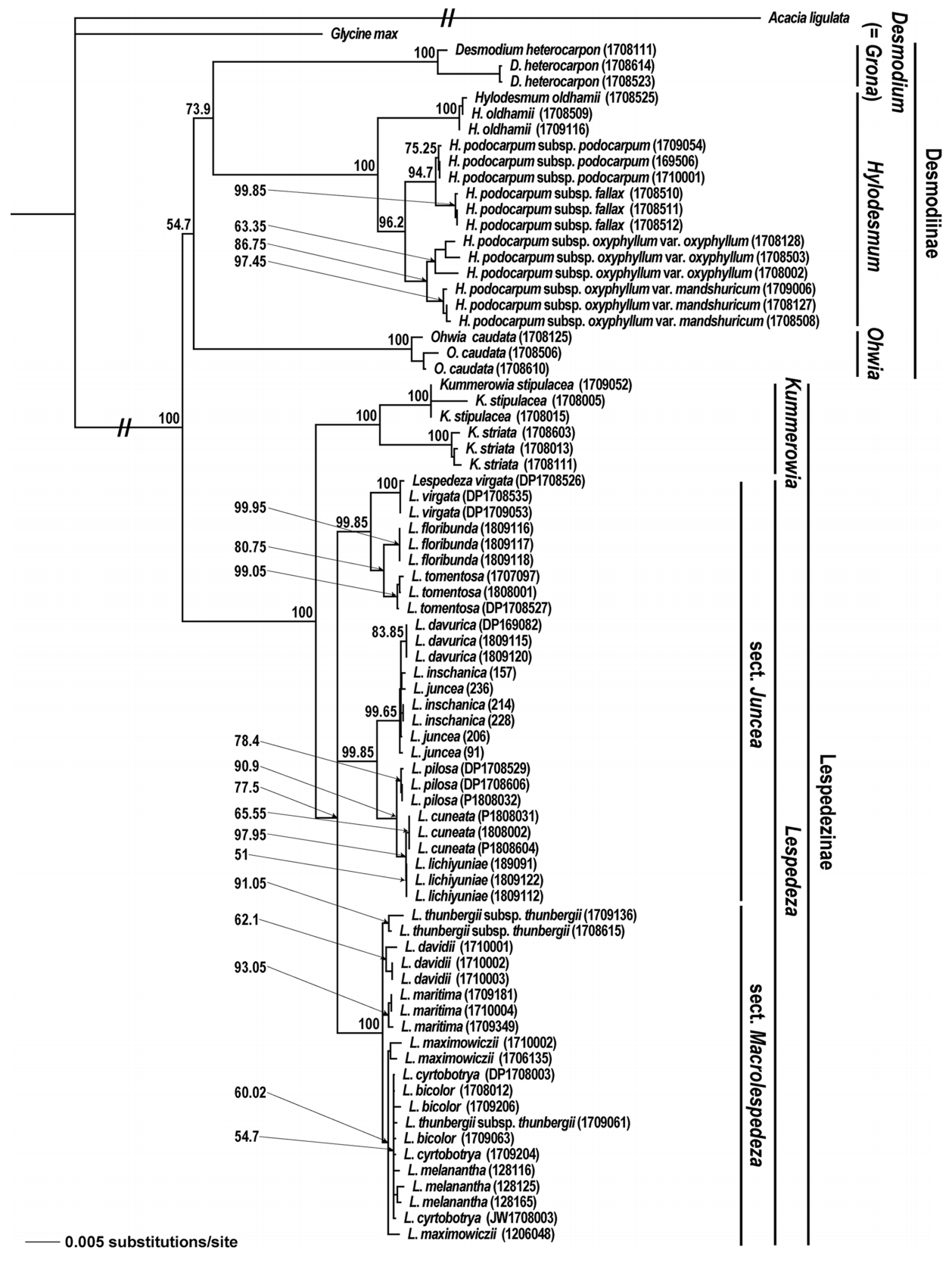
Table 1.
Statistics of three DNA barcode loci of tribe Desmodieae in Korea.
|
rbcL |
matK |
ITS |
Total |
|
No. of taxa (accessions) successfully analyzed |
25 (75) |
25 (75) |
25 (75) |
25 (75) |
|
Sequencing success (%) |
100 |
100 |
100 |
100 |
|
Sequence length (bp) |
715 |
813–822 |
594–648 |
2,125–2,176 |
|
Aligned length (bp) |
715 |
832 |
746 |
2293 |
|
G + C ratio (%) |
42.2 |
27.6 |
61.6 |
42.1 |
|
No. of variable sites (% variable sites) |
23 (3.22) |
108 (12.98) |
100 (13.40) |
231 (10.07) |
Table 2.
Summary of the pairwise intraspecific and interspecific distances in the DNA barcode loci of tribe Desmodieae species.
|
Region |
Intraspecific K2P distance
|
Interspecific K2P distance
|
|
Min. |
Mean |
Max. |
SD |
Min. |
Mean |
Max. |
SD |
|
rbcL |
0 |
0.0002 |
0.0028 |
0.0006 |
0 |
0.0192 |
0.0463 |
0.0183 |
|
matK |
0 |
0.0019 |
0.0275 |
0.0050 |
0 |
0.0179 |
0.0640 |
0.0170 |
|
ITS |
0 |
0.0026 |
0.0205 |
0.0048 |
0 |
0.1099 |
0.2216 |
0.0637 |
|
All combined |
0 |
0.0015 |
0.0109 |
0.0022 |
0 |
0.0424 |
0.0944 |
0.0283 |
Table 3.
Species identification rate of tribe Desmodieae in Korea using the DNA barcode.
|
Regions |
Species identification rate (%) |
|
rbcL |
28 |
|
matK |
20 |
|
ITS |
68 |
|
rbcL + matK |
32 |
|
rbcL + ITS |
68 |
|
matK + ITS |
64 |
|
rbcL + matK + ITS |
72 |
Literature Cited
Akiyama, S.. 1988. A revision of the genus Lespedeza section Macrolespedeza (Leguminosae). The University Museum, The University of Tokyo, Bulletin 33: 1-170.
CBOL Plant Working Group. 2009. A DNA barcode for land plants. Proceedings of the National Academy of Sciences of the United States of America 106: 12794-12797.   Choi, B.-H.. 1991. Morphological variation and taxonomy of Desmodium podocarpium (Leguminosae) in southern Korea. Korean Journal of Plant Taxonomy 21: 55-69 (in Korean).   Choi, B.H.. 2007. Lespedeza Michx. In The Genera of Vascular Plants of Korea. Park, C.-W. (ed.), Academy Publishing Co, Seoul. 614-618.
Choi, Y.G.. Youm, J.W.. Lim, C.E. and Oh, H.. 2018. Phylogenetic analysis of Viburnum (Adoxaceae) in Korea using DNA sequences. Korean Journal of Plant Taxonomy 48: 206-217.   Chung, K.-A. and Cheong, M.-J.. 2016. Effects of Lespedeza Caneata ethanol extract on the liver, kidneys of lead administered mice. Journal of the Korea Academia-Industrial cooperation Society 17: 207-214 (in Korean).   Dong, W.. Liu, J.. Yu, J.. Wang, L. and Zhou, S.. 2012. Highly variable chloroplast markers for evaluating plant phylogeny at low taxonomic levels and for DNA barcoding. PLoS ONE 7: e35071.   Dong, W.. Xu, C.. Li, C.. Sun, J.. Zuo, Y.. Shi, S.. Cheng, T.. Guo, J. and Zhou, S.. 2015. ycf1, the most promising plastid DNA barcode of land plants. Scientific Reports 5: 8348.     Edwards, D.. Horn, A.. Taylor, D.. Savolainen, V. and Hawkins, J.A.. 2008. DNA barcoding of a large genus, Aspalathus L. (Fabaceae). Taxon 57: 1317-1327.  Eun, J.S.. 2011. Effect of Lespedeza cuneata G. Don on the activity of murine immune cells. Korean Journal of Oriental Physiology Pathology 25: 837-842 (in Korean).
Gao, T.. Yao, H.. Song, J.. Liu, C.. Zhu, Y.. Ma, X.. Pang, X.. Xu, H. and Chen, S.. 2010. Identification of medicinal plants in the family Fabaceae using a potential DNA barcode ITS2. Journal of Ethnopharmacology 130: 116-121.   Han, J.E. and Choi, B.-H.. 2008. Two naturalized plants of Lespedeza (Leguminosae) in Korea: L. lichiyuniae & L. davidii. Korean Journal of Plant Taxonomy 38: 547-555 (in Korean).  Han, J.E.. Chung, K.-H.. Nemoto, T. and Choi, B.-H.. 2010. Phylogenetic analysis of eastern Asian and eastern North American disjunct Lespedeza (Fabaceae) inferred from nuclear ribosomal ITS and plastid region sequences. Botanical Journal of the Linnean Society 164: 221-235.  Hasebe, M.. Omori, T.. Nakazawa, M.. Sano, T.. Kato, M. and Iwatsuki, K.. 1994. rbcL gene sequences provide evidence for the evolutionary lineages of leptosporangiate ferns. Proceedings of the National Academy of Sciences of the United States of America 91: 5730-5734.    Hatusima, S.. 1967. Lespedeza: sects. Macrolespedeza and Heterolespedeza from Japan, Corea and Formosa. Memoirs of the Faculty of Agriculture, Kagoshima University 6: 1-17.
Hebert, P.D.N.. Cywinska, A.. Ball, S.L. and deWaard, J.R.. 2003. Biological identifications through DNA barcodes. Proceedings of the Royal Society of London Series B Biological Sciences 270: 313-321.   Hollingsworth, P.M.. Graham, S.W. and Little, D.P.. 2011. Choosing and using a plant DNA barcode. PLoS ONE 6: e19254.   Huang, P.H. and Ohashi, H.. 2010. Desmodium Desvaux. In Flora of China. Vol. 10. Wu, Z. Y.. Raven, P. H.. Hong, D. Y. (eds.), Science Press, Beijing and Missouri Botanical Garden Press, St. Louis, MO. 268-278.
Huang, P.H.. Ohashi, H. and Nemoto, T.. 2010. Lespedeza Michaux. unknown, .. Ohashi, H.. Nemoto, T. (eds.), Science Press, Beijing and Missouri Botanical Garden Press. 302-311.
Jabbour, F.. Gaudeul, M.. Lambourdière, J.. Ramstein, G.. Hassanin, A.. Labat, J.-N. and Sarthou, C.. 2018. Phylogeny, biogeography and character evolution in the tribe Desmodieae (Fabaceae: Papilionoideae), with special emphasis on the New Caledonian endemic genera. Molecular Phylogenetics and Evolution 118: 108-121.   Kajita, T. and Ohashi, H.. 1994. Chloroplast DNA variation in Desmodium subgenus Podocarpium (Leguminosae): infrageneric phylogeny and infraspecific variations. Journal of Plant Research 107: 349-354.   Kajita, T.. Ohashi, H.. Tateishi, Y.. Bailey, C.D. and Doyle, J.J.. 2001. rbcL and legume phylogeny, with particular reference to Phaseoleae, Millettieae, and allies. Systematic Botany 26: 515-536.
Kim, S.M.. Kang, K.. Jho, E.H.. Jung, Y.-J.. Nho, C.W.. Um, B.-H. and Pan, C.-H.. 2011. Hepatoprotective effect of flavonoid glycosides from Lespedeza cuneata against oxidative stress induced by tert?butyl hyperoxide. Phytotherapy Research 25: 1011-1017.  Kimura, M.. 1980. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. Journal of Molecular Evolution 16: 111-120.   Kress, W.J.. Wurdack, K.J.. Zimmer, E.A.. Weigt, L.A. and Janzen, D.H.. 2005. Use of DNA barcodes to identify flowering plants. Proceedings of the National Academy of Sciences of the United States of America 102: 8369-8374.   Kress, W.J. and Erickson, D.L.. 2008. DNA barcodes: genes, genomics, and bioinformatics. Proceedings of the National Academy of Sciences of the United States of America 105: 2761-2762.   Lee, J.-Y.. Kim, M.J.. Oh, C.-K. and Choi, B.-H.. 2012. First record of Hylodesmum laxum (Fabaceae) from Korea. Korean Journal of Plant Taxonomy 42: 207-210.
Legume Phylogeny Working Group. 2013. Legume phylogeny and classification in the 21st century: progress, prospects and lessons for other species-rich clades. Taxon 62: 217-248.
Legume Phylogeny Working Group. 2017. A new subfamily classification of the Leguminosae based on a taxonomically comprehensive phylogeny. Taxon 66: 44-77.
Lee, T.B.. 1965. The Lespedeza of Korea (1). Bulletin of the Seoul National University Forests 2: 1-43.
Lee, T.B.. 1980. Illustrated Flora of Korea. Hyangmunsa, Seoul. 468-472.
Li, H.-C.. Zhao, L.. Gao, F. and Xu, B.. 2019. Molecular phylogeny of the genus Hylodesmum (Fabaceae). Phytotaxa 403: 221-229.
Meusnier, I.. Singer, G.A.C.. Landry, J.-F.. Hickey, D.A.. Hebert, P. D. N. and Hajibabaei, M.. 2008. A universal DNA mini-barcode for biodiversity analysis. BMC Genomics 9: 214.   Meyer, C.P. and Paulay, G.. 2005. DNA barcoding: error rates based on comprehensive sampling. PLoS Biology 3: e422.   Nakai, T.. 1927. Lespedeza of Japan and Korea. The Forestal Experiment Station of Government General of Chosen 6: 1-101.
Nemoto, T.. Yokoyama, J.. Fukuda, T.. Iokawa, Y. and Ohashi, H.. 2010. Phylogeny of Lespedeza (Leguminosae) based on chloroplast trnL-trnF sequences. Journal of Japanese Botany 85: 213-229.
Ohashi, H.. 1973. The Asiatic species of Desmodium and its allied genera (Leguminosae). Ginkgoana 1: 142-155.
Ohashi, H.. 1999. The genera, tribes and subfamilies of Japanese Leguminosae. The Science Reports of the Tohoku University, 4th Series. Biology 40: 187-268.
Ohashi, H.. 2005. Desmodieae. In Legumes of the World. Lewis, G.. Schrire, B.. Mackinder, B.. Lock, M. (eds.), Royal Botanic Gardens, Kew. 432-445.
Ohashi, H.. Polhill, R.H. and Schubert, B.G.. 1981. Desmodieae. In Advances in Legume Systematics. Part 1. Polhill, R. M.. Raven, P. H. (eds.), Royal Botanic Gardens, Kew. 292-300.
Ohashi, H. and Mill, R.R.. 2000. Hylodesmum, a new name for Podocarpium (Leguminosae). Edinburgh Journal of Botany 57: 171-188.  Ohashi, H. and Nemoto, T.. 2014. A new system of Lespedeza (Leguminosae tribe Desmodieae). Journal of Japanese Botany 89: 1-11.
Ohashi, H. and Ohashi, K.. 2012. Ototropis, a genus separated from Desmodium (Leguminosae). Journal of Japanese Botany 87: 108-118.
Ohashi, H. and Ohashi, K.. 2018a. Grona, a genus separated from Desmodium (Leguminosae tribe Desmodieae). Journal of Japanese Botany 93: 104-120.
Ohashi, H. and Ohashi, K.. 2018b. Sohmaea, a new genus of Leguminosae tribe Desmodieae. Journal of Japanese Botany 93: 104-120.
Ohashi, K.. Ohashi, H.. Nemoto, T.. Ikeda, T.. Izumi, H.. Kobayashi, H.. Muragaki, H.. Nata, K.. Sato, N. and Suzuki, M.. 2018a. Phylogenetic analyses for a new classification of the Desmodium group of Leguminosae tribe Desmodieae. Journal of Japanese Botany 93: 165-189.
Ohashi, K.. Ohashi, H.. Nemoto, T.. Abe, C.. Kotani, H.. Nata, K.. Ohtake, H. and Yamamoto, K.. 2018b. Phylogenetic analyses for classification of the Desmodium group of Leguminosae tribe Desmodieae 2. Two new genera separated from Desmodium and two new combinations in Grona and Sohmaea. Journal of Japanese Botany 93: 293-306.
Purty, R.S. and Chatterjee, S.. 2016. DNA barcoding: an effective technique in molecular taxonomy. Austin Journal of Biotechnology & Bioengineering 3: 1059.
Renner, S.S.. 1999. Circumscription and phylogeny of the Laurales: evidence from molecular and morphological data. American Journal of Botany 86: 1301-1315.   Riahi, M.. Zarre, S.. Maassoumi, A.A.. Osaloo, S.K. and Wojciechowski, M.F.. 2011. Towards a phylogeny for Astragalus section Caprini (Fabaceae) and its allies based on nuclear and plastid DNA sequences. Plant Systematics and Evolution 293: 119.   Salazar, G.A.. Chase, M.W.. Soto Arenas, M.A. and Ingrouille, M.. 2003. Phylogenetics of Cranichideae with emphasis on Spiranthinae (Orchidaceae, Orchidoideae): evidence from plastid and nuclear DNA sequences. American Journal of Botany 90: 777-795.   Schindler, A.K.. 1916. Desmodiinae novae. Botanische Jahrbücher für Systematik, Pflanzengeschichte und Pflanzengeographie 54: 51-68.
Swofford, D. L.. 2002. PAUP 4.0b10: Phylogenetic analysis using parsimony. Sinauer Associates, Sunderland, MA.
Vassou, S.L.. Kusuma, G. and Parani, M.. 2015. DNA barcoding for species identification from dried and powdered plant parts: a case study with authentication of the raw drug market samples of Sida cordifolia. Gene 559: 86-93.   White, T.J.. Bruns, T.. Lee, S. and Taylor, J.. 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In PCR Protocols: A Guide to Methods and Applications. Innis, M. A.. Gelfand, D. H.. Sninsky, J. J.. White, T. J. (eds.), Academic Press, New York. 315-322.
Wojciechowski, M.F.. Lavin, M. and Sanderson, M.J.. 2004. A phylogeny of legumes (Leguminosae) based on analysis of the plastid matK gene resolves many well?supported subclades within the family. American Journal of Botany 91: 1846-1862.   Xu, B.. Wu, N.. Gao, F. and Zhang, B.. 2012. Analysis of DNA sequences of six chloroplast and nuclear genes suggests incongruence, introgression, and incomplete lineage sorting in the evolution of Lespedeza (Fabaceae). Molecular Phylogenetics and Evolution 62: 346-358.   Youm, J.W.. Han, W.. Seo, S.W.. Lim, C.U. and Oh, H.. 2016. DNA barcoding of Schisandraceae in Korea. Korean Journal of Plant Taxonomy 46: 273-282.  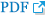
APPENDICES
Appendix 1.
List of species sequenced in this study.
|
Locality (deposition of voucher specimen) |
Voucher No. |
Collector |
Date |
|
Ohwia caudata (된장풀) |
|
Dongbaekdongsan, Jocheon-eup, Jeju-si, Jeju-do (KB) |
1708506 |
D. P. Jin & J. S. Park |
23 Aug 2017 |
|
Seogwipo Healing Forest, Hogeun-dong, Seogwipo-si, Jeju-do (KB) |
1708610 |
D. P. Jin & J. S. Park |
24 Aug 2017 |
|
Yeongcheonak volcanic cone, Sanghyo-dong, Seogwipo-si, Jeju-do (KB) |
1708125 |
I. S. Choi & J. W. Park |
24 Aug 2017 |
|
Desmodium heterocarpon (= Grona heterocarpa) (잔디갈고리) |
|
Hogeun cemetery, Hogeun-dong, Seogwipo-si, Jeju-do (KB) |
1708111 |
I. S. Choi & J. W. Park |
24 Aug 2017 |
|
Sallok road, Harye-ri, Seogwipo-si, Jeju-do (KB) |
1708523 |
D. P. Jin & J. S. Park |
23 Aug 2017 |
|
Seogwipo Healing Forest, Hogeun-dong, Seogwipo-si, Jeju-do (KB) |
1708614 |
D. P. Jin & J. S. Park |
24 Aug 2017 |
|
Hylodesmum oldhamii (큰도둑놈의갈고리) |
|
Sallok road, Harye-ri, Seogwipo-si, Jeju-do (KB) |
1708525 |
D. P. Jin & J. S. Park |
23 Aug 2017 |
|
Bijarim forest, Pyeongdae-ri, Jeju-si, Jeju-do (KB) |
1708509 |
D. P. Jin & J. S. Park |
23 Aug 2017 |
|
Gayasan Mt., Hapcheon-gun, Gyeongsangnam-do (KB) |
1709116 |
J. W. Park |
8 Sep 2017 |
|
H. podocarpum subsp. fallax (긴도둑놈의갈고리) |
|
Bijarim forest, Pyeongdae-ri, Jeju-si, Jeju-do (KB) |
1708510–1708512 |
D. P. Jin & J. S. Park |
23 Aug 2017 |
|
H. podocarpum subsp. oxyphyllum var. mandshuricum (애기도둑놈의갈고리) |
|
Jeoksangsan Mt., Muju-gun, Jeollabuk-do (KB) |
1709006 |
D. P. Jin |
7 Sep 2017 |
|
Bijarim forest, Pyeongdae-ri, Jeju-si, Jeju-do (KB) |
1708508 |
D. P. Jin & J. S. Park |
23 Aug 2017 |
|
Georinsaseum, Daepo-dong, Seogwipo-si, Jeju-do (KB) |
1708127 |
I. S. Choi & J. W. Park |
24 Aug 2017 |
|
H. podocarpum subsp. oxyphyllum var. oxyphyllum (도둑놈의갈고리) |
|
Geomdansan Mt., Hanam-si, Gyeonggi-do (KB) |
1708002 |
D. P. Jin & J. W. Park |
4 Aug 2017 |
|
Yeongcheonak volcanic cone, Sanghyo-dong, Seogwipo-si, Jeju-do (KB) |
1708128 |
I. S. Choi & J. W. Park |
24 Aug 2017 |
|
Dongbaekdongsan, Jocheon-eup, Jeju-si, Jeju-do (KB) |
1708503 |
D. P. Jin & J. S. Park |
23 Aug 2017 |
|
H. podocarpum subsp. podocarpum (개도둑놈의갈고리) |
|
Gujinsan Mt., Changnyeong-gun, Gyeongsangnam-do (KB) |
1709054 |
D. P. Jin |
8 Sep 2017 |
|
Oeyeondo Isl., Boryeong-si, Chungcheongnam-do (KB) |
169506 |
D. P. Jin |
30 Sep 2016 |
|
Unbongsan Mt., Boryeong-si, Chungcheongnam-do (IUI) |
1710001 |
J. S. Park |
21 Oct 2017 |
|
Kummerowia stipulacea (둥근매듭풀) |
|
Gujinsan Mt., Changnyeong-gun, Gyeongsangnam-do (KB) |
1709052 |
D. P. Jin |
8 Sep 2017 |
|
Buldang-ri, Gwangju-si, Gyeonggi-do (KB) |
1708015 |
D. P. Jin & J. W. Park |
4 Aug 2017 |
|
Geomdansan Mt., Hanam-si, Gyeonggi-do (KB) |
1708005 |
D. P. Jin & J. W. Park |
4 Aug 2017 |
|
K. striata (매듭풀) |
|
Seogwipo Healing Forest, Hogeun-dong, Seogwipo-si, Jeju-do (KB) |
1708608 |
D. P. Jin & J. S. Park |
24 Aug 2017 |
|
Buldang-ri, Gwangju-si, Gyeonggi-do (KB) |
1708013 |
D. P. Jin & J. W. Park |
4 Aug 2017 |
|
Seonyasan Mt., Geumsan-gun, Chungcheongnam-do (KB) |
1708111 |
J. W. Park |
21 Aug 2017 |
|
Lespedeza bicolor (싸리) |
|
Hwanghaksan Mt., Chilgok-gun, Gyeongsangbuk-do (KB) |
1709063 |
D. P. Jin |
9 Sep 2017 |
|
Buldang-ri, Gwangju-si, Gyeonggi-do (KB) |
1708012 |
D. P. Jin & J. W. Park |
4 Aug 2017 |
|
Biseulsan Mt., Cheongdo-gun, Gyeongsangbuk-do (KB) |
1709206 |
J. W. Park |
15 Sep 201 |
|
L. cyrtobotrya (참싸리) |
|
Sikjangsan Mt., Dong-gu, Daejeon-si (KB) |
JW1708003 |
J. W. Park |
6 Aug 2017 |
|
Namhansanseong Fortress, Gwangju-si, Gyeonggi-do (KB) |
DP1708003 |
D. P. Jin & J. W. Park |
4 Aug 2017 |
|
Biseulsan Mt., Cheongdo-gun, Gyeongsangbuk-do (KB) |
1709204 |
J. W. Park |
15 Sep 2017 |
|
L. davidii (큰잎싸리) |
|
Agricultural Research and Extension Service, Hwangdeung-myeon, Iksan-si, Jeollabuk-do (KB) |
1710001–1710003 |
D. P. Jin |
7 Oct 2017 |
|
L. maritima (해변싸리) |
|
Paljoryeong Pass, Cheongdo-gun, Gyeongsangbuk-do (KB) |
1709181 |
D. P. Jin |
16 Sep 2017 |
|
Bogildo Isl., Wando-gun, Jeollanam-do (KB) |
1710004 |
J. W. Park |
15 Oct 2017 |
|
Gujinsan Mt., Changnyeong-gun, Gyeongsangnam-do (KB) |
1709349 |
D. P. Jin |
8 Sep 2017 |
|
L. maximowiczii (조록싸리) |
|
Goseongsan Mt., Gimcheon-si, Gyeongsangbuk-do (KB) |
1706135 |
D. P. Jin |
25 May 2017 |
|
Cheongwansan Mt., Jangheung-gun, Jeollanam-do (KB) |
1710002 |
J. W. Park |
14 Oct 2017 |
|
Jeoksangsan Mt., Muju-gun, Jeollabuk-do (KB) |
1206048 |
D. P. Jin |
22 Jun 2012 |
|
L. melanantha (검나무싸리) |
|
Nogodan, Jirisan Mt., Gurye-gun, Jeollanam-do (KB) |
128116 |
D. P. Jin |
13 Aug 2013 |
|
Banyabong, Jirisan Mt., Namwon-si, Jeollabuk-do (KB) |
128125 |
D. P. Jin |
13 Aug 2013 |
|
Gayasan Mt., Hapcheon-gun, Gyeongsangnam-do (KB) |
128165 |
D. P. Jin |
14 Aug 2013 |
|
L. thunbergii subsp. thunbergii (풀싸리) |
|
Hwanghaksan Mt., Chilgok-gun, Gyeongsangbuk-do (KB) |
1709061 |
D. P. Jin |
9 Sep 2017 |
|
Seogwipo Healing Forest, Seogwipo-si, Jeju-do (KB) |
1708615 |
D. P. Jin & J. S. Park |
24 Aug 2017 |
|
Paljoryeong Pass, Cheongdo-gun, Gyeongsangbuk-do (KB) |
1709136 |
D. P. Jin |
16 Sep 2017 |
|
L. cuneata (비수리) |
|
Daedae-ri, Ulju-gun, Ulsan-si (KB) |
P1808031 |
J. W. Park |
3 Aug 2018 |
|
Sorae Marsh Ecological Park, Namdong-gu, Incheon-si (KB) |
1808002 |
J. S. Park |
10 Aug 2018 |
|
Wondang-dong, Samcheok-si, Gangwon-do (KB) |
P1808604 |
J. W. Park |
31 Aug 2018 |
|
L. davurica (호비수리) |
|
Hagoe-ri, Danyang-gun, Chungcheongbuk-do (KB) |
DP169082 |
D. P. Jin |
10 Sep 2016 |
|
Haneul Park, Mapo-gu, Seoul-si (KB) |
1809115 |
J. S. Park |
21 Sep 2018 |
|
Masian Beach, Jung-gu, Incheon-si (KB) |
1809120 |
J. S. Park |
21 Sep 2018 |
|
L. floribunda (분홍싸리) |
|
Haneul Park, Mapo-gu, Seoul-si (KB) |
1809116–1809118 |
J. S. Park |
21 Sep 2018 |
|
L. inschanica (청비수리) |
|
Sangha Reservoir, Seongmodo Isl., Ganghwa-gun, Incheon-si (IUI) |
157 |
J. E. Han |
5 Sep 2004 |
|
Waryongsan Mt., Sacheon-si, Gyeongsangnam-do (IUI) |
214 |
J. E. Han |
3 Sep 2006 |
|
Sinduri Beach, Taean-gun, Chungcheongnam-do (IUI) |
228 |
J. E. Han |
21 Sep 2004 |
|
L. juncea (땅비수리) |
|
Wangsan Beach, Jung-gu, Incheon-si (IUI) |
91 |
J. E. Han |
1 Sep 2004 |
|
Gwanggyosan Mt., Yongin-si, Gyeonggi-do (IUI) |
206 |
J. E. Han |
17 Sep 2004 |
|
Sinduri Beach, Taean-gun, Chungcheongnam-do (IUI) |
236 |
J. E. Han |
21 Sep 2004 |
|
L. lichiyuniae (자주비수리) |
|
Chilbosan Mt., Hwaseong-si, Gyeonggi-do (KB) |
DP189091 |
D. P. Jin |
14 Sep 2018 |
|
Insam Land Rest Area, Geumsan-gun, Chungcheongnam-do (KB) |
1809122 |
J. S. Park |
15 Sep 2018 |
|
Haneul Park, Mapo-gu, Seoul-si (KB) |
1809112 |
J. S. Park |
21 Sep 2018 |
|
L. pilosa (괭이싸리) |
|
Sallok road, Harye-ri, Seogwipo-si, Jeju-do (KB) |
DP1708529 |
D. P. Jin & J. S. Park |
23 Aug 2017 |
|
Seogwipo Healing Forest, Hogeun-dong, Seogwipo-si, Jeju-do (KB) |
DP1708606 |
D. P. Jin & J. S. Park |
24 Aug 2017 |
|
Daedae-ri, Ungchon-myeon, Ulju-gun, Ulsan-si (KB) |
P1808032 |
J. W. Park |
3 Aug 2018 |
|
L. tomentosa (개싸리) |
|
Seongmodo Isl., Ganghwa-gun, Incheon-si (KB) |
1707097 |
I. S. Choi |
14 Jul 2017 |
|
Sallok road, Harye-ri, Seogwipo-si, Jeju-do (KB) |
DP1708527 |
D. P. Jin & J. S. Park |
23 Aug 2017 |
|
Sorae Marsh Ecological Park, Namdong-gu, Incheon-si (KB) |
1808001 |
J. S. Park |
10 Aug 2018 |
|
L. virgata (좀싸리) |
|
Sallok road, Harye-ri, Seogwipo-si, Jeju-do (KB) |
DP1708526, DP1708535 |
D. P. Jin & J. S. Park |
23 Aug 2017 |
|
Gujinsan Mt., Changnyeong-gun, Gyeongsangnam-do (KB) |
DP1709053 |
D. P. Jin |
8 Sep 2017 |
Appendix 2.
The List of primer used in the present study.
|
Region |
Primer name |
Primer sequence (5'-3') |
Reference |
|
ITS |
ITS5 |
F GGA AGT AAA AGT CGT AAC AAG G |
White et al. (1990) |
|
ITS4 |
R TCC TCC GCT TAT TGA TAT GC |
|
|
matK |
matK4La |
F CCT TCG ATA CTG GGT GAA AGA T |
Wojciechoeski et al. (2004) |
|
matK1932Ra |
R CCA GAC CGG CTT ACT AAT GGG |
|
|
matK1100L |
F TTC AGT GGT ACG GAG TCA AAT G |
|
|
trnK2R |
R CCC GGA ACT AGT CGG ATG G |
|
|
FGA309F |
F AGT ACT TTT GTG CTT ACG AGC C |
Newly designed |
|
FGA309R |
R ATT ACT ATT GTG ACG GGA ACA G |
|
|
rbcL |
rbcL_aF |
F ATG TCA CCA CAA ACA GAG ACT AAA GC |
Hasebe et al. (1994) |
|
rbcL_afR |
R CAT GCA TTA CGA TAG GAA CGC C |
Newly designed |
|
|















